Liberando o nó de enfermidades xenéticas
2017/03/01 Garcia Etxebarria, Koldo - Genetikan doktoreaEHUko Genetika, Antropologia Fisikoa eta Animalia Fisiologia saila Iturria: Elhuyar aldizkaria

A base de moitas enfermidades, preto de 14.000, atópase en defectos xenómicos. Estas enfermidades poden clasificarse en dous grandes grupos: por unha banda, cando un único xene provócao, denomínanse enfermidades monogénicas ou mendelianas; e por outro, cando interveñen varios xenes, as denominadas enfermidades xenéticas complexas. O primeiro paso é descubrir cales son os xenes relacionados coas enfermidades, que é imprescindible paira atopar remedio. A pesar de que nos últimos anos producíronse grandes avances no coñecemento destes xenes defectuosos, a solución non está tan clara.
As enfermidades causadas polos únicos xenes, unhas 4.000, foron as primeiras en analizarse. Ao seguir os patróns que predín as leis de Mendel, é máis fácil atopar un xene relacionado coa enfermidade. Paira iso analízase a distribución da enfermidade nunha familia e clasifícase en función dela.
Cando se produce por un xene que se atopa nun dos 22 cromosomas non sexuais, chámase enfermidade autosómica. Nos autosómicos dominantes, a única copia defectuosa do xene é suficiente paira desenvolver a enfermidade. Entre elas, as máis frecuentes son a hipercolesterolemia familiar (una de cada 500 recentemente nados), a neurofibromatosis tipo I (una de cada 2.500) ou a enfermidade de Huntington (una de cada 15.000).
Nas enfermidades autosómicas recesivas, as dúas copias do xene deben ser defectuosas paira o desenvolvemento da enfermidade. A anemia falciforme (una de cada 625), a fibrosis quística (una de 2.000) e a enfermidade de Tay-Sachs (una de cada 3.000) son as enfermidades máis estendidas deste tipo.
En canto aos cromosomas sexuais, entre as enfermidades relacionadas cos xenes situados no cromosoma X atópanse a distrofia muscular de Duchenne (una de cada 7.000) e a hemofilia (una de cada 10.000). No caso do cromosoma E, con todo, son moi raros e normalmente provocan esterilidad.
Aínda que puxemos como exemplo as enfermidades monogénicas máis estendidas, a maioría das enfermidades mendelianas son enfermidades raras, é dicir, menos dunha de cada 2.000 persoas sófrenas. Con todo, coñécese o xene responsable. Cando se sospeita que un paciente padece una enfermidade, pódese secuenciar ese xene concreto e confirmar si ten esa mutación coñecida (cambio de secuencia). Con todo, pode ocorrer que non haxa esa mutación coñecida, é dicir, que ese paciente teña una nova mutación. Paira buscar esta nova mutación son moi útiles as técnicas de secuenciación de todo o xenoma que se desenvolveron nos últimos anos.
Na actualidade somos capaces de obter a secuencia da rexión funcional de todos os xenes (uns 20.000) dunha persoa (o que se coñece como exoma) e de analizar as mutacións que estes levan. É habitual que haxa certos cambios, xa que o material xenético de todos os seres humanos non é exactamente o mesmo; ademais, a maioría dos cambios non interferen na función do xene. Pero comparando o exoma do enfermo co dos seus pais pódese deducir que cambios recibiu dos seus pais e cales son novos, e cales deles poden ter efectos adversos sobre a función do xene.
Doutra banda, as complexas enfermidades xenéticas están máis estendidas na poboación, pero non é tan fácil atopar os xenes responsables das mesmas. Enfermidades como a asma, a esclerose múltiple ou a diabetes. Ademais, a pesar do seu compoñente xenético, debido á influencia do medio, o estudo da enfermidade é máis complicado.
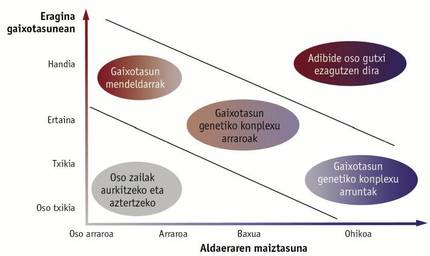
Paira identificar os xenes implicados utilizouse a técnica coñecida como análise asociativa de todo o xenoma. Mediante esta técnica analízanse os marcadores xenéticos SNP (do inglés single nucleotide polymorphism, polimorfismo mononucleótido) dispersos por todo o xenoma. Desenvolvéronse ferramentas paira analizar simultaneamente miles de marcadores deste tipo, coñecidos como microarray, o que permite coñecer de forma áxil e sinxela a variedade de SNPs que leva cada persoa analizada. Se una variante de SNP é máis frecuente en persoas con enfermidade que en persoas sas, pódese sospeitar que esta variante está relacionada coa enfermidade. Realizáronse preto de 2.400 estudos deste tipo, que asociaron de forma significativa case 17.000 SNPs a unha enfermidade ou característica complexa (Figura 1).
Con todo, non somos capaces de identificar todos os xenes asociados a enfermidades xenéticas complexas. Sabemos que hai poucos xenes que inflúen na enfermidade, pero estes non son suficientes paira explicar completamente o desenvolvemento da enfermidade. Moitas enfermidades xenéticas complexas son consecuencia da combinación de moitos SNP comúns, que elevan ou reducen lixeiramente o risco de padecer una enfermidade (Figura 2). Ademais, este grupo de SNP non ten por que estar nun xene, é dicir, non ten por que crear un erro nun xene. Isto dificulta a interpretación de que uno ou varios SNP están relacionados cunha enfermidade. Pode ser que estes SNPs atópense preto de una mutación que realmente produce un erro e non teñan un efecto real. Tamén pode ocorrer que o SNP inflúa na regulación do xene e que, aínda que o xene non presente defectos, non estea á altura necesaria. Aínda queda moito por facer paira coñecer a orixe xenética de enfermidades xenéticas complexas, e todo iso será un reto paira o futuro inmediato.
Una vez que sexamos capaces de establecer a orixe dunha enfermidade xenética, o seguinte reto sería atopar o seu remedio. Até o momento, as únicas solucións exitosas foron a mitigación dos efectos da enfermidade ou a prevención das condicións ambientais que a poden producir. De feito, a reparación directa dos defectos génicos, a terapia xenética, non foi tan exitosa como quixésemos até agora, polo que non se xeneralizou.
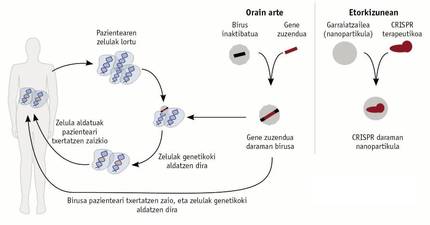
Naqueles casos nos que un xene determinado non é capaz de cumprir a súa función, realizáronse ensaios paira incorporar un xene que funcione correctamente no material xenético dalgúns pacientes (Figura 3). Paira iso introdúcese un xene funcional nun virus modificado, que é administrado ao paciente como tratamento e integrado no material xenético das células do paciente. Esta estratexia comezou a aplicarse nalgúns pacientes cunha grave inmunodeficiencia combinada en 1990, pero a súa eficacia era a curto prazo. Buscando una solución a longo prazo, mellorouse a técnica e no ano 2000 realizáronse novos ensaios. Desgraciadamente, como consecuencia marxinal destes novos ensaios, os pacientes desenvolveron una leucemia que provocou o seu abandono. Este efecto colateral tiña a súa orixe en que o virus introducía a copia funcional do xene noutro xene, o que xeraba leucemia.
Desde 2015 parece que vivimos una resurrección das terapias génicas. Técnicas como o CRISPR, os dedos de zinc son capaces de evitar limitacións e problemas asociados ao uso de virus (Figura 3). Estas técnicas son capaces de coñecer o xene que se quere resolver, polo que teñen una especificidad que os virus non teñen. Estas técnicas xa están a dar moito que dicir e a curto prazo van dar aínda máis. Deberá comprobarse a súa eficacia.
O desenvolvemento de ferramentas paira coñecer os fundamentos das enfermidades xenéticas permitiunos comprender moito mellor estas enfermidades e parece que empezamos a imaxinar remedios eficaces. Aínda que con cautela, podemos ser optimistas e pensar que nun futuro próximo imos vivir una revolución ao redor das enfermidades xenéticas.
Bibliografía

Gai honi buruzko eduki gehiago
Elhuyarrek garatutako teknologia