Doble hélice medio siglo
¿Cuál es la razón para conocer la maquinaria de la vida? ¿Curiosidad? ¿Control? ¿Poder crear nuevos seres vivos? ¿Cuáles son las principales causas de curación? ¿Sufrir lo menos posible? ¿Alargar la vida? ¿Por qué la larga vida es mejor que la corta? ¿Por qué huimos de la muerte? ¿Por miedo? ¿Hasta qué punto el ser humano tiene derecho a transformar el ciclo de la vida?
Estas preguntas han salido de los orígenes de la biología. Quizás antes. Sin embargo, la investigación del ADN ha resucitado y actualizado. Pero no es una cuenta instantánea. Para ver de dónde viene esa actualización basta con tirar de la investigación. ¿Hasta cuándo? Al menos hasta que el microscopio se convirtió en una herramienta habitual para los biólogos.

Las células eran muy interesantes por muchas razones. Por ejemplo, guardan el secreto de la herencia: todas las células son copias de otras células anteriores. Las células crean nuevas células. ¿Cómo es posible? El microscopio no respondía. Realizaron muchos experimentos para buscar respuesta. Y poco a poco los resultados orientaron la investigación hacia el núcleo celular.
Sin embargo, la herencia no es sólo copiar células. Además, a través de la herencia, las características del ser que se reproduce se repiten en el ser que nace. A partir de esta idea, en 1906, el biólogo de Wilhem Ludwig Johannsen inventó la palabra gene. El gen era un "algo" que transmite una característica, pero XX. A principios del siglo XX los científicos no tenían ni idea de cómo era ese 'algo' y de qué está hecho.
¿Cuál es el objetivo?
Si se observa bien, dentro del núcleo de las células se ve una mezcla de materia. Y si atrapamos a la célula mientras se copia, se ve claramente que el núcleo de la confusión son los filamentos. En 1888, el alemán Wilhem von Waldeyer propuso la palabra cromosoma para designar los filamentos.
Waldeyer no pudo ver el aspecto de estos filamentos; hoy sabemos cómo separar los componentes de esta mezcla y que los cromosomas tienen forma de X. Pero para la mayoría de los científicos de la época, el aspecto de los cromosomas no era importante. Por el contrario, querían saber si los cromosomas y los genes eran lo mismo.
Los cromosomas y los genes no son lo mismo. Sin embargo, hay razones para pensar lo contrario. En definitiva, tanto los cromosomas como los genes están 'repetidos' en cada célula. ¿Por qué no será un cromosoma la información de una característica del ser? Walter S. El citólogo Sutton tenía muy claro que el hombre heredaba miles de cualidades y sólo tiene 23 cromosomas diferentes. No había duda. En cada cromosoma se necesitaban muchos genes. Entonces, ¿cómo son los genes físicamente?
Bombas y bacterias
La estructura de la molécula de ADN es la respuesta a esta pregunta. Ahora lo sabemos. Pero hacia 1920 los científicos no lo tenían nada claro. En el núcleo, junto con el ADN, había proteínas, pero no se sabía cuál era su función.
¿ADN o proteínas? ¿Quién determina las características del ser vivo? Sin ningún otro tipo de análisis, las proteínas parecen moléculas mucho más complejas e interesantes que el ADN.

En definitiva, las partes básicas necesarias para formar una proteína tienen una variedad mucho mayor que las necesarias para formar ADN. Hay veinte aminoácidos que pueden intervenir en la estructura de las proteínas, mientras que el ADN utiliza sólo cuatro nucleótidos. (Esto hace que la estructura del ADN no varíe de una molécula a otra y que los cambios entre proteínas sean enormes. Sin embargo, los biólogos desconocían entonces la estructura de estas moléculas). Sin embargo, en la década de 1930 no era posible responder a esta pregunta. No es de extrañar.
Para investigar lo que está dentro del núcleo se necesitan muchos recursos, y en esa época los científicos no disponían de los equipos adecuados y no sabían los conceptos básicos que había que saber. Quizá era demasiado pronto para la biología. Entre otras cosas, necesitaba física. Por ejemplo, hay que recordar que la difracción de rayos X fue fundamental para el estudio de las estructuras de las biomoléculas.
Predominaba la física en la ciencia. Así lo piensan los historiadores de la ciencia. El átomo, la radiación electromagnética, la relatividad, la mecánica cuántica… estaban emergiendo nuevos conceptos físicos que alteraron la ciencia. La primera mitad del siglo fue para la física, la ciencia era innovadora para una sociedad innovadora y estaba dispuesta, entre otras cosas, a aplicarla en guerras totalmente innovadoras. Tras la explosión de la bomba atómica en Hiroshima, muchos científicos se asustaron con la capacidad del átomo, pero ya no se podía retroceder.
Estructura y premio
Pero la guerra no fue estéril desde el punto de vista biológico. En 1944, los estadounidenses Oswald Avery, Colin McLeod y Maclyn McCarty dieron un paso importante: Vieron que el ADN era la clave de la herencia.
Estos biólogos trabajaban con los neumáticos. En el laboratorio se empleaban dos tipos de neumococos, uno con una superficie de rugosidad y otro con cubierta superficial suave. En el experimento de los científicos se extrae el ADN de unos "suaves" del neumococo muerto y se inyecta a los arrugados vivos. Con el tiempo, estos últimos desarrollaron una cubierta lisa, es decir, aunque provengan de organismos muertos, el ADN podía transmitir características.
Este descubrimiento impulsó la investigación del ADN. Además, al finalizar la guerra, los investigadores europeos mejoraron notablemente sus condiciones de trabajo. En esta situación, la investigación del ADN era muy tentadora, ya que al tratarse de una molécula tan importante, probablemente el que clarificaba la estructura ganaría el premio Nobel.
En esta investigación participaron los ingleses James Watson, Francis Crick, Maurice Wilkins y Rosalind Franklin. Es una historia oscura que ha sido tema de muchos escritos (por ejemplo, véase Elhuyar Zientzia eta Teknika 185, 20-23). pág. ).
El 28 de febrero de 1953 Watson y Crick propusieron el modelo de doble hélice, que fue publicado en el número 25 de abril de la revista Nature junto con otras investigaciones. Por este descubrimiento fueron galardonados con el Premio Nobel de Medicina de 1962 junto a Maurice Wilkins. Rosalind Franklin ya había muerto.
ADN, estructura elegante
Han cumplido cincuenta años desde que Watson y Crick dieron a conocer la estructura del ADN y el mundo ha querido celebrarlo. Estos días se escuchará con frecuencia la definición de la estructura del ADN. que ha sido uno de los hitos científicos del siglo XX. Sin embargo, ese mismo año, en 1953, Max Perutz sentó las bases para aclarar la estructura de las proteínas; y en 1955, Sanger leyó por primera vez la secuencia de aminoácidos de una proteína.
¿Tienen menos importancia que la doble hélice? Está claro que no. Entonces, ¿por qué tanta desequilibrio en las celebraciones? Parece que la clave está en la elegancia.
La doble hélice es una estructura elegante que se ha convertido en el icono de la ciencia moderna. Los artistas también lo quieren. Pero hace 50 años no fue así.
En los años siguientes al descubrimiento la doble hélice no recibió gran atención por parte de los científicos. En las revistas Science y Nature hasta 1960, por ejemplo, pocos artículos que hablaban de ADN hicieron referencia a la estructura de Watson y Crick. Curioso porque entonces se investigaba mucho sobre el ADN.
Sin embargo, la relación entre el ADN y las proteínas no estaba muy clara y, sobre todo, no sabían que las órdenes de elaboración estaban almacenadas en el ADN. Para Robert Olby, esta es la razón por la que se dio tan pocos casos a la estructura del ADN. Olby Pittsburg, profesor emérito de Filosofía e Historia de la Ciencia de la Universidad, ha recogido los datos mencionados.
A principios de la década de 1950 se sabía que el ADN era la molécula de la herencia. También sabían que está junto con las proteínas en el núcleo, pero no todas las piezas estaban unidas. Todavía no entendieron que los indicadores de las características que pasan de generación en generación son proteínas. Por ello, el ADN y las proteínas se estudiaban en dos líneas de investigación diferentes.

El gran paso hacia la unificación de líneas lo dio el astrónomo George Gamow. En los últimos años de su vida comenzó a investigar el ADN y propuso una teoría sobre cómo el ADN codificaba la información. En su opinión, este código estaba formado por órdenes de elaboración de proteínas. ¿Pero cuál era el código concreto?
Lingüistas del ADN
El ADN estaba compuesto por cuatro letras que debían traducirse a otro idioma de 20 letras, el de las proteínas. ¿Cuál era la combinación correcta? Las letras del ADN tomadas de dos en dos eran dieciséis opciones, por lo tanto insuficientes, y de tres en 64. Podría ser. Pero, si era así, ¿qué trío correspondía a cada aminoácido? ¿Sólo valía uno o más?
La propuesta de Gamow suscitó gran interés entre los científicos y creó un club con veinte científicos para fomentar la comunicación y la amistad y proponer y discutir posibles códigos: Club de corbata RNA. Era un club especial. Cada miembro tenía una corbata con la estructura del ARN y una aguja con la abreviatura del nombre de un aminoácido. Esta abreviatura era el sobrenombre de su compañero. En este club participaron, entre otros, Watson y Crick. Era 1954 y las cosas empiezan a cambiar. Muchos amantes de las proteínas se dedicaron más a mirar el ADN.
Tres años después, en 1957, Crick en el Simposio de la Asociación de Biología Experimental dio a conocer el “Dogma principal”: Del ADN al ARN, a la proteína. Cuatro años después comprendieron las primeras letras del lenguaje genético.
En un libro reconoció que utilizó la palabra dogma para llamar la atención, sin saber qué quería decir realmente. El dogma es una afirmación que se considera una verdad básica, sea o no pruebas. Pero en este caso no se puede decir que no se haya probado la de Crick. Hoy todos creemos en ello.
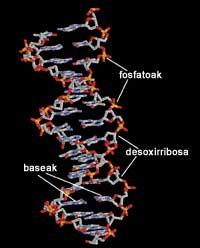
Las hélices son características de la biología, son estructuras estables por lo que su función de soporte es sencilla. Sin embargo, el caso del ADN es especial: es una doble hélice que no cumple funciones estructurales; es el refugio de la información.
Las dos hélices están enrolladas entre sí, en dirección contraria, es decir, colocando "arriba" la secuencia repetitiva de las unidades de los azúcares de fosfato de un filamento, el del otro filamento queda ordenado "abajo".
Es una estructura muy elegante, sencilla y eficaz: las unidades azucareras desoxiribosa y los grupos de fosfatos se encuentran en el exterior de la hélice, mientras que en el interior se encuentran las bases para mantener unidos ambos filamentos. La información del ADN se basa en la secuencia de estas bases.
Las células humanas tienen 23 pares de cromosomas, la mitad de padres y la mitad de madre. Los óvulos y espermatozoides sólo 23. Y estos cromosomas no son puros. De una célula de 23 pares de cromosomas se forman dos óvulos, pero antes de su distribución se produce el proceso denominado recombinación. Entre pares de cromosomas con la misma información, las partes de ADN se intercambian, de alguna manera se mezcla la información del padre y de la madre y luego se produce la división. Así, de generación en generación no se transmite la misma información genética. En 1972 se consiguió hacer lo mismo en el laboratorio. Investigadores de la Universidad de Standford crearon una molécula de ADN de material genético de ambos virus. En 1970 se identificaron en las bacterias enzimas capaces de conocer y cortar determinadas secuencias de la cadena de ADN y, dos años después, Paul Berg aisló a una de ellas para cortar el ADN. Cortó el ADN del virus del mono SV40 que produce cáncer y lo unificó con un virus que contamina las bacterias a través de otras enzimas. El siguiente paso es esa molécula de ADN E. Coli era injertado en la bacteria, pero quien lo consiguió se asustó o dejó ahí el experimento. Ese mismo año, junto con otros investigadores, publicó una carta en la revista Science, en la que propone suspender durante un año las investigaciones sobre la recombinación del ADN. Pero no tuvo éxito. El primer organismo modificado genéticamente nació en la Universidad de Stanford en 1973. |
La información que contiene el material genético se puede leer 'manualmente', pero la bacteria más pequeña también tiene muchos datos. La cantidad de datos es un problema grave tanto en la recogida, como en el almacenamiento y en el análisis.
La solución a este problema llegó en 1986. El estadounidense Leroy Hood desarrolló la primera máquina que lee automáticamente la secuencia del ADN. Esto supuso un gran impulso a los proyectos creados para conocer los genes.
El mayor eco de estos proyectos está relacionado con la secuenciación completa del genoma humano. Se trata de un gigantesco proyecto de acceso y análisis de la información contenida en los 23 cromosomas del ser humano, supuestamente finalizado.

La metodología que comenzó a utilizar el PNS era lenta y la primera idea era finalizar el trabajo durante quince años, aunque los avances permitieron reducir el plazo y anunciaron que finalizaría en 2003.
En 1998 supieron que una empresa privada puso en marcha el mismo proyecto, la empresa Celera Genomics Corporation. Esta empresa quería utilizar una metodología nueva y más rápida. Entonces, los PNB cambiaron la estrategia intentando acelerar de alguna manera el proceso. En lugar de realizar el trabajo con gran precisión, en un plazo más breve se elaborará un borrador del mapa del genoma. A partir de este borrador se acometería un trabajo concreto.
La empresa Celera Genomics tenía otra estrategia: romper todo el genoma y leer el código de todos los trozos. Antes de sumergirse totalmente en el proyecto, Celera Genomics confirmó que la metodología era más rápida utilizando genomas de pequeños organismos.
El 13 de febrero de 2001, las revistas científicas más prestigiosas del mundo, Nature y Science, publicaron sendos números especiales para la presentación de dos borradores del genoma humano, de la organización del Proyecto Internacional de Genoma Humano y de la empresa privada Celera Genomics, respectivamente.

Buletina
Bidali zure helbide elektronikoa eta jaso asteroko buletina zure sarrera-ontzian








